Chemical Bonds - HyperPhysics
hyperphysics.phy-astr.gsu.edu/hbase/chemical/bond.html
翻譯這個網頁
翻譯這個網頁
Covalent chemical bonds involve the sharing of a pair of valence electrons by two atoms, in contrast to the transfer of electrons in ionic bonds. Such bonds lead
In the book, Schrödinger introduced the idea of an "aperiodic crystal" that contained genetic information in its configuration of covalent chemical bonds. In the 1950s, this idea stimulated enthusiasm for discovering the genetic molecule. Although the existence of DNA had been known since 1869, its role in reproduction and its helical shape were still unknown at the time of Schrödinger's lecture. In retrospect, Schrödinger's aperiodic crystal can be viewed as a well-reasoned theoretical prediction of what biologists should have been looking for during their search for genetic material. Both James D. Watson,[2] and independently, Francis Crick, co-discoverers of the structure of DNA, credited Schrödinger's book with presenting an early theoretical description of how the storage of genetic information would work, and each respectively acknowledged the book as a source of inspiration for their initial researches.[3]
蛋白质结构测定的未来
|
马上就要毕业了,我的方向是生物大分子结构,所以写了一个蛋白质结构测定的未来。最初写这个,其实就是想向身边的人解释一下我在做什么以及我打算做什么,所以用的语言很通俗,例子也是大家熟悉的,可是后来因为功力不够的原因,不免写得复杂了。
简单地说明一下我的背景,在北京高能所读凝聚态专业的博士,在北京同步辐射装置生物大分子线站值过班,同时在结构生物实验室(后来叫蛋白质中心)干过活,主要工作内容是用同步辐射方法研究红细胞和血红蛋白,目标是想看看细胞内血红蛋白的状态。
虽然题目很大,其实就是我对于蛋白质结构的一些理解,也没有新东西,如果不是这个方向的也许看了会了解一二。是这个方向的前辈,对于错误的地方请多多指教。
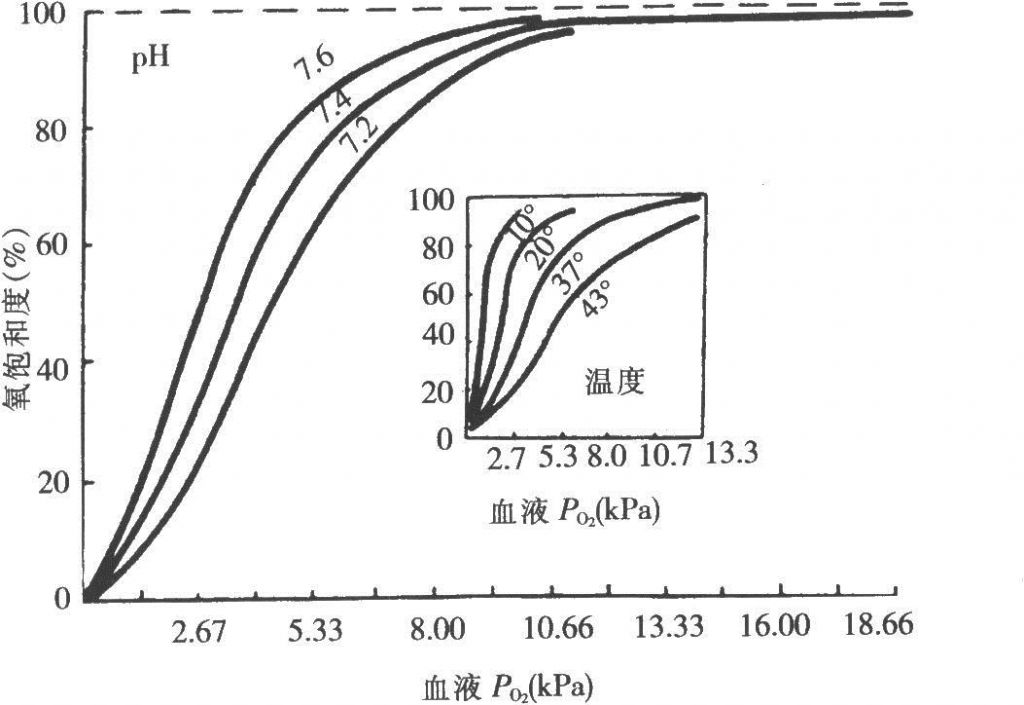
1、蛋白质结构及其与功能的关系 既然要说未来,就要说一下现状。蛋白质是生命活动的载体。机体中的每一个细胞和所有重要组成部分都有蛋白质参与,生命活动最终都要靠蛋白质实现。举个例子,呼吸活动。我们每天都在吸入氧气,排出二氧化碳。正是氧气帮助我们把吃进去的食物变成我们进行活动的能量。通过气管吸入肺中的氧气要通过血液输运到身体组织中,这里面就有一种蛋白的功劳:血红蛋白。氧气结合到血红蛋白上,到组织的时候,就将氧释放出来,被组织吸收!同时将二氧化碳运走。见图1,这是血红蛋白的氧解离曲线,如果拿血液做实验,得到的结果也是一致的,这是因为真正起作用的是血红蛋白。图中随着温度和pH值的变化,氧结合的比例会有所不同,正是这种细微的调节,可以决定身体不同部位接受氧的量。
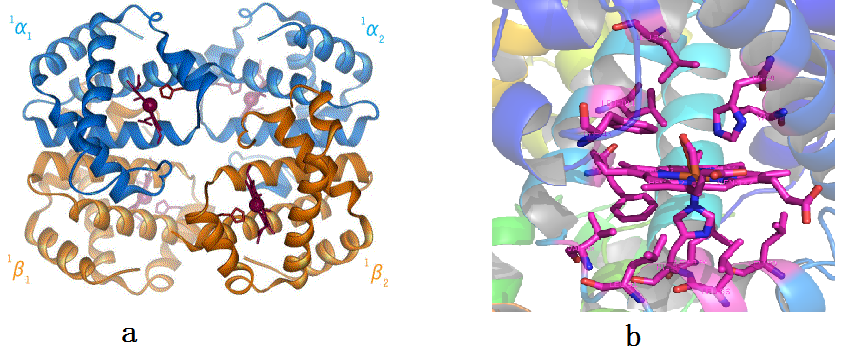
前面只是说明蛋白质很重要,那么跟结构有什么关系呢?蛋白质的结构决定了它功能的实现。我们看细胞,里面包含有无数个蛋白质。其实是有一个大致的数的,人的红细胞直径通常是6μm~8μm,血红蛋白的直径约为6nm,细胞内32%的体积为血红蛋白,就算按红细胞最小直径算,又因为红细胞是扁平的,也可以知道一个细胞内至少有一亿个血红蛋白。这里面的问题就是一个细胞中可能有成百上千种蛋白,而且每一种都有大量的拷贝,它们是如何稳定共存的呢?答案是在稳定的溶液环境中,蛋白有一个稳定的结构。这里面还是先举血红蛋白为例子,见图2。图中a为血红蛋白的整体结构,由四个相似的部分(亚基)构成,这些亚基之间的相对位置会发生变化,引起氧结合能力的变化。图b显示的氧结合部位的放大结构,氧就结合在血红素平面上。
现在人们已经测定了将近十万个蛋白质结构。广为人知的事实是,蛋白质的基本组成单元并不是原子,而是氨基酸。就如我们现在砌房子不会直接用泥土,而是用砖块,这样更省事。在氨基酸之上,几个或者十几个氨基酸会形成稳定的二级结构,比如图2a中的螺旋。这些结构通过一定的方式折叠才能形成稳定的三级结构,有了生物活性,就是前面所说的亚基。有些蛋白是由多个亚基构成的,这些亚基通过氢键、疏水作用相互连接,一起完成更重要的功能。但我们不要忘了,所有的一切都是由原子构成的。
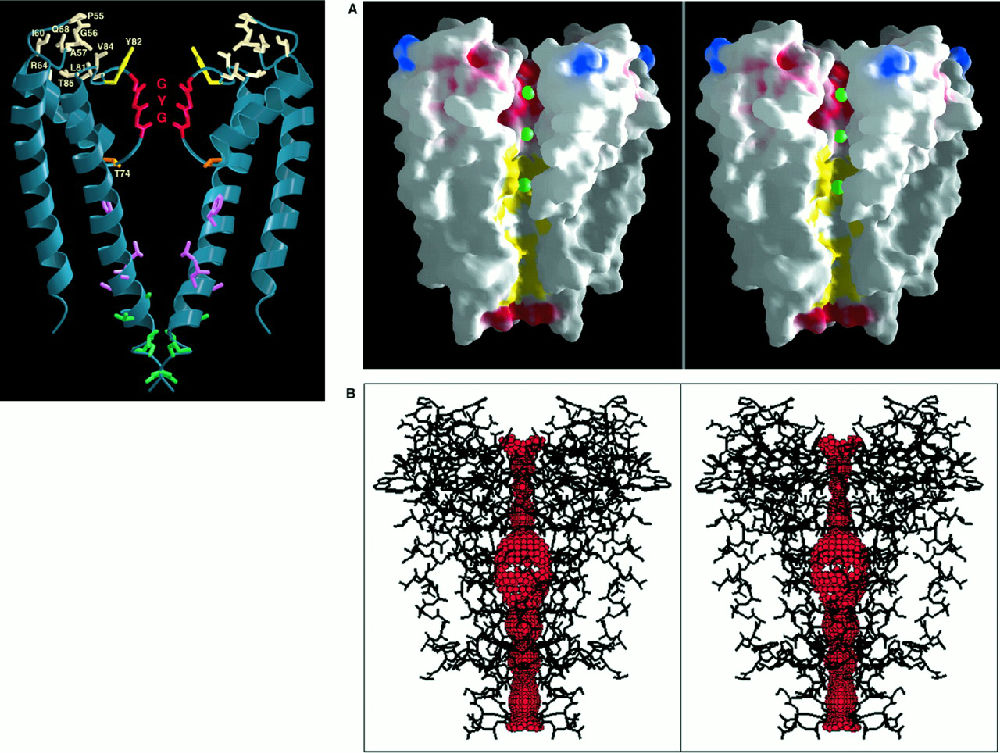
在这里再举一个结构的例子。1998年,R. Mackinnon解出了离子通道的结构,然后获得了2003年的诺贝尔奖。一个多世纪前,Wilhelm Ostwald首先提出,生物体内的电信号是离子进出细胞膜而产生的。到了1950年代初, Alan Hodgkin和Andrew Huxley发现,这些离子是钾离子和钠离子。这样的叙事只是交待了一个时间线,神经信号是一种电信号,这种信号是利用细胞内的外的离子浓度差造成的电势差形成的,但是这种离子浓度差并不能用简单的渗透压和扩散来解释,需要有一种机器通过消耗能量使钾离子进入细胞内,同时将钠离子排出。这样的装置还要在一定的电势作用下开启,以维持神经信号的传递。这里另外一个重要事实是,神经信号在细胞内的传播是不衰减的。Mackinnon发现的就是这样一种机器的结构(见图3)。四个类似的亚基组成一个桶状结构插入细胞膜中,中间留有一个通道供离子进出,图A表明一次进入的是3个钾离子。图B将离子通道突出了出来。问题在于钾离子比钠离子大,为什么钾离子能进来,钠离子进不去呢?离子在溶液中存在并不是单个原子的形式,而是由水包裹的,离子通道能够脱去钾离子的水“外衣”,即左侧的GYG结构,但是对钠离子没有办法。这个通道的大小是0.27nm,钾离子的大小。
到这里简单说明了蛋白质的结构以及结构对于揭示功能的重要性,下面要说的就是现在测定蛋白质结构的方法,前面的结论也是来自于这些方法。然后会分析一下这些方法的优劣,最后是面临的问题和突破方向。
2、结构测定的方法 有个数据库就叫蛋白质结构数据库(protein data bank,PDB),表1统计了它收集的蛋白质结构数目,使用的方法。可以看到主要的方法是X射线,其次是核磁共振(NMR),以及电镜(EM),第四种是联合使用多种方法,最后一项其他是中子散射。这里主要介绍前三种方法,而且以第一种为主。为什么?同步辐射就是一种X射线光源,第二它最主流,因为相对其他两种方法,它有十分明显的优势。
X射线其实是X射线晶体学的简写,一般使用的波长是1埃左右,也就是0.1nm。这个尺度刚好也是原子半径大小。
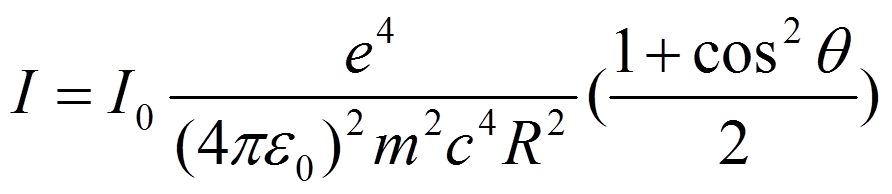 。
。
需要注意的跟质量的平方成反比,质子的质量是电子的1800倍,所以电子的散射强度比质子强得多,我们得到的信号主要来自于电子的散射。另外就是跟散射角度的关系,角度越高,散射越弱。当有多个电子的时候,电子之间的位置关系用来表示,波矢用来表示,散射出来的波就有一个相位关系,用电子密度来表示电子的位置,就有
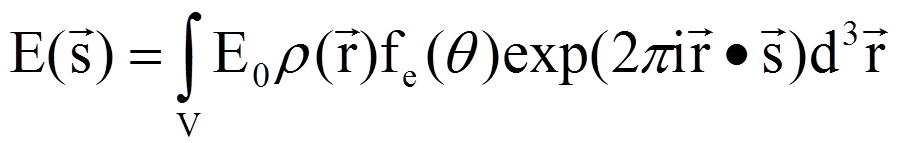
电镜的全称是电子显微镜。使用的是电子作为探针,用电磁透镜代替了光学透镜并使用荧光屏将肉眼不可见电子束成像。为什么不使用透镜来让x光成像呢?原因在于硬X射线的穿透性,目前还无法做X射线透镜。
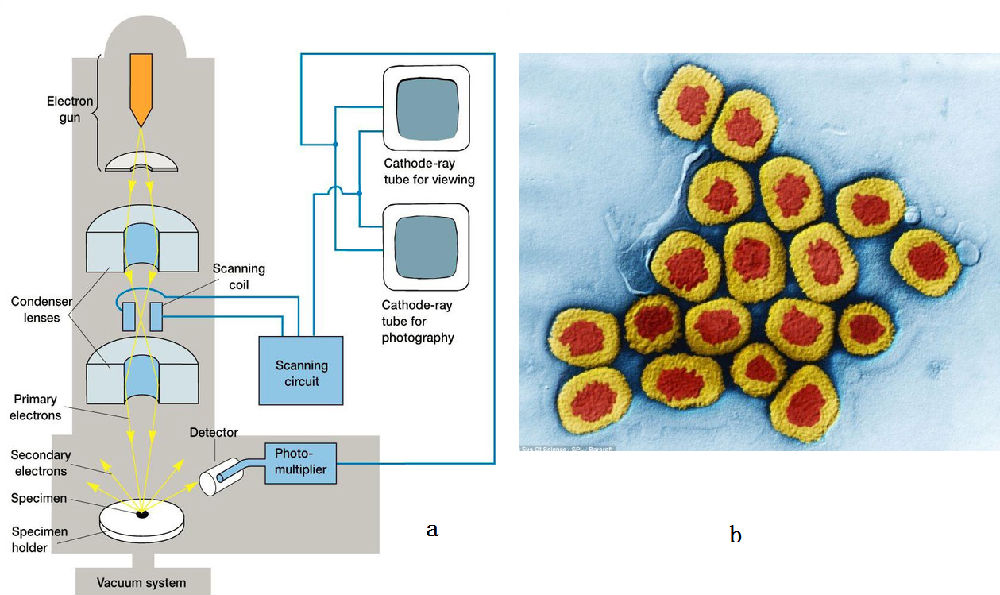
电子打在样品上,会激发出二次电子、或X射线等,通过探测二次电子可以得到当前位置的电子强度信息。然后移到附近,这样就可以得到图7b中的图像。图中的颜色是假彩色,真实的信息是二次电子的强弱。按照成像理论,这样得到的只是一个样品的截面,如果有许多全同的样品,扫描得到不同截面的信息,就可以利用计算机技术将这些截面组合起来,得到样品的三维结构。
这里面存在的问题是,电子的散射很强,很容易被空气散射,所以需要高真空环境。这样一来样品就不能是活的。由于技术原因,目前最好的分辨率不到3埃,这和晶体学相比是有差距的。
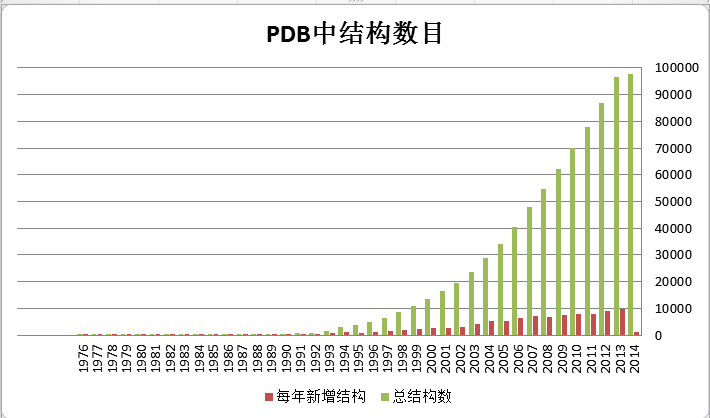
3、蛋白质结构测定的未来 前面大致介绍了一下蛋白质结构测定的常用方法。总体来说,这几种方法越来越成熟,每年测得的结构也越来越多。下表是截至2014年2月PDB数据库中的结构数目,可以看到结构数目呈指数增长。国内而言,也有越来越多的生物学家进入结构测定这一领域,因为结构确实能解释他们研究中的很多问题,加深对于生物意义的理解。
单纯就结构测定而言,能解释的问题是有限。一个蛋白长成一个样子,然后我们说这样就可以跟DNA结合,可以作为离子通道,可以跟某个小分子结合,这些在生物学实验中可能就已经得到验证了。给你一个蛋白结构,如果对于其在细胞中的定位以及功能了解很少的话,其实是很难讲一个好故事的。这就是生物学家做结构的优势。他们可能在细胞或者生化层面做研究,到了分子层面,以前的那些疑问可能都会得到解答。他原来就知道蛋白有某种特性,必须与DNA结合,拿到结构一看,蛋白质的凹槽正好可以结合DNA。某种疾病引起了基因突变,他把正常的和突变的蛋白结构一比较,就可以发现问题出现在什么地方。这是结构测定的一大前景。
前面已经说过电镜和NMR的问题,晶体学的最大瓶颈在于结晶。结晶的方法是得到高纯度的蛋白,然后将蛋白加入各种配好的溶液中进行结晶。具体哪个溶液能够长出晶体是不知道的,只能不停地去尝试。而且已经测得的结构如此之多,那些容易结晶的蛋白质可能已经被尝试过了,现在的方向是复合物。由好几个蛋白质通过相互作用构成的复合物一起实现一个功能,但是和单个蛋白相比,复合物之间的相互作用比较弱,这样对溶液环境的要求就更高。也就是说更难找到结晶条件了,而且就算是长成晶体,由于复合物结构复杂,很难是完美晶体,衍射的分辨率就上不去。
回到结构本身。在小分子领域,量子化学的计算是相当精确的。但在蛋白质结构中,由于原子众多,用量化计算的话计算量非常大,所以常用的分子动力学。通过构置分子力场,就是分子中原子相互作用规则,然后利用最优化方法,得到一个“能量”最小的结构。而为了兼顾精度和计算速度,人们会使用QM/MM这样的组合,即在核心区域使用量化进行计算,而在外围使用分子力学。蛋白质中的原子是作为一个整体存在的,配体结合在活性位点会影响整个蛋白的结构。很多蛋白使用ATP作为动力来源,ATP末端磷酸基团水解时,释放出的能量是30.54 kJ/mol。这样的能量够离子泵运作一次。显然,你结晶的时候是无法把ATP水解的过程固定住,NMR测定一个结构的时间要几天。而一个电子在蛋白质中的转移可能就在ps量级。这很好理解,一个反应中,中间态是不稳定,只有初态和末态才能大量存在,就如血红蛋白中结合氧和没有结合氧,你很难找到一个即将结合氧的结构。但是我们可以自己制造。
时间分辨的晶体学是在飞秒化学的基础上发展起来的。利用飞秒激光作为时间基准,可以在反应之后的特定时间进行测量,这样就可能得到反应中间态的信息。比如肌红蛋白结合氧的实验,但是氧和肌红蛋白结合的能力不如CO(CO中毒的原因就在于此,结合到血红蛋白之后不容易解离,造成氧输运受阻),所以一般这类实验采用的是结合CO的肌红蛋白,使用特定频率的光照,可以使CO脱离蛋白中的铁原子,脱离之后CO是不是飞出结合的口袋呢?可以在解离之后特定的时间去测量红外光谱,这样响应时间很快。而要直观地看到结构,则使用X射线,在150ps内,CO还是停留在肌红蛋白的口袋中,大约在1ms之内会重新结合到铁原子上。为了更快地进行测量,往往使用“白光”,这样的衍射叫劳厄衍射,一张衍射图就可能解出结构。显然这样的实验也存在问题,比如使用晶体,这样光解实验变化的区域只能活性位点附近。
如果有一种方法能够得到单分子的结构,那么就可以很方便地得到中间态。这就是X射线自由电子激光最诱人的应用。X射线自由电子激光(FEL)的基本原理是通过自由电子和辐射的相互作用,电子将能量传送给辐射而使辐射强度放大。由直线加速器产生的电子束流,经过几次压缩后将能产生超短的脉冲长度(fs量级),将为研究物质的微观动态结构以及非线性过程提供前所未有的机会。
图8是利用FEL进行单颗粒成像的示意图。蛋白颗粒从一个质谱仪中喷出,一束相干X光照射上去,由于光束其高的能量,蛋白被击碎。但是在被击碎之前,已经将X射线散射出来,里面含有蛋白结构信息。这仍然是电子密度的傅里叶变换,可以反向重建蛋白质的结构。现在的问题是一张图的信号太差,需要许多张一样的图平均来增强信号,得到的仍然是结构的一个截面,这与电镜类似,所以需要多张图来重建结构。最大的问题是,高角度的散射信号非常弱,不到一个光子,于是分辨率也上不去。
使用更强的光源,更短的脉冲,以及更好的探测器,是否有可能使用一张图就解出结构来呢?如果答案是能的话,那么要多强的光,多短的脉冲,多好的探测器?现在美国斯坦福的LCLS已经为用户进行供光,位于德国汉堡的X-FEL和日本的SCSS都在建设之中。相关的实验已经进行了几十个,但对于单张图成像方法和理论都需要更多的探索。
写到这里,其实很多东西没有写明白,只是给出一个大致的轮廓。在单分子测量领域,光镊和磁镊对分子力学进行了很多有意义的测量;荧光能量共振转移(FRET)已经成为检测活体中生物大分子纳米级距离和纳米级距离变化的有力工具,在生物大分子相互作用时间分析有着广泛的应用;而生物学家在生物大分子相互作用也有很多非常具有创造性的方法。总体来看,时间分辨的大分子结构测定只是我们对于分子的理解需要的更多信息的一种可能,但是是最直观的一种!

http://blog.sciencenet.cn/blog-502850-767838.html 此文来自科学网刘广峰博客,转载请注明出处。
简单地说明一下我的背景,在北京高能所读凝聚态专业的博士,在北京同步辐射装置生物大分子线站值过班,同时在结构生物实验室(后来叫蛋白质中心)干过活,主要工作内容是用同步辐射方法研究红细胞和血红蛋白,目标是想看看细胞内血红蛋白的状态。
虽然题目很大,其实就是我对于蛋白质结构的一些理解,也没有新东西,如果不是这个方向的也许看了会了解一二。是这个方向的前辈,对于错误的地方请多多指教。
1、蛋白质结构及其与功能的关系 既然要说未来,就要说一下现状。蛋白质是生命活动的载体。机体中的每一个细胞和所有重要组成部分都有蛋白质参与,生命活动最终都要靠蛋白质实现。举个例子,呼吸活动。我们每天都在吸入氧气,排出二氧化碳。正是氧气帮助我们把吃进去的食物变成我们进行活动的能量。通过气管吸入肺中的氧气要通过血液输运到身体组织中,这里面就有一种蛋白的功劳:血红蛋白。氧气结合到血红蛋白上,到组织的时候,就将氧释放出来,被组织吸收!同时将二氧化碳运走。见图1,这是血红蛋白的氧解离曲线,如果拿血液做实验,得到的结果也是一致的,这是因为真正起作用的是血红蛋白。图中随着温度和pH值的变化,氧结合的比例会有所不同,正是这种细微的调节,可以决定身体不同部位接受氧的量。

图1,血红蛋白的氧解离曲线
前面只是说明蛋白质很重要,那么跟结构有什么关系呢?蛋白质的结构决定了它功能的实现。我们看细胞,里面包含有无数个蛋白质。其实是有一个大致的数的,人的红细胞直径通常是6μm~8μm,血红蛋白的直径约为6nm,细胞内32%的体积为血红蛋白,就算按红细胞最小直径算,又因为红细胞是扁平的,也可以知道一个细胞内至少有一亿个血红蛋白。这里面的问题就是一个细胞中可能有成百上千种蛋白,而且每一种都有大量的拷贝,它们是如何稳定共存的呢?答案是在稳定的溶液环境中,蛋白有一个稳定的结构。这里面还是先举血红蛋白为例子,见图2。图中a为血红蛋白的整体结构,由四个相似的部分(亚基)构成,这些亚基之间的相对位置会发生变化,引起氧结合能力的变化。图b显示的氧结合部位的放大结构,氧就结合在血红素平面上。

图2,血红蛋白的结构
现在人们已经测定了将近十万个蛋白质结构。广为人知的事实是,蛋白质的基本组成单元并不是原子,而是氨基酸。就如我们现在砌房子不会直接用泥土,而是用砖块,这样更省事。在氨基酸之上,几个或者十几个氨基酸会形成稳定的二级结构,比如图2a中的螺旋。这些结构通过一定的方式折叠才能形成稳定的三级结构,有了生物活性,就是前面所说的亚基。有些蛋白是由多个亚基构成的,这些亚基通过氢键、疏水作用相互连接,一起完成更重要的功能。但我们不要忘了,所有的一切都是由原子构成的。
在这里再举一个结构的例子。1998年,R. Mackinnon解出了离子通道的结构,然后获得了2003年的诺贝尔奖。一个多世纪前,Wilhelm Ostwald首先提出,生物体内的电信号是离子进出细胞膜而产生的。到了1950年代初, Alan Hodgkin和Andrew Huxley发现,这些离子是钾离子和钠离子。这样的叙事只是交待了一个时间线,神经信号是一种电信号,这种信号是利用细胞内的外的离子浓度差造成的电势差形成的,但是这种离子浓度差并不能用简单的渗透压和扩散来解释,需要有一种机器通过消耗能量使钾离子进入细胞内,同时将钠离子排出。这样的装置还要在一定的电势作用下开启,以维持神经信号的传递。这里另外一个重要事实是,神经信号在细胞内的传播是不衰减的。Mackinnon发现的就是这样一种机器的结构(见图3)。四个类似的亚基组成一个桶状结构插入细胞膜中,中间留有一个通道供离子进出,图A表明一次进入的是3个钾离子。图B将离子通道突出了出来。问题在于钾离子比钠离子大,为什么钾离子能进来,钠离子进不去呢?离子在溶液中存在并不是单个原子的形式,而是由水包裹的,离子通道能够脱去钾离子的水“外衣”,即左侧的GYG结构,但是对钠离子没有办法。这个通道的大小是0.27nm,钾离子的大小。
到这里简单说明了蛋白质的结构以及结构对于揭示功能的重要性,下面要说的就是现在测定蛋白质结构的方法,前面的结论也是来自于这些方法。然后会分析一下这些方法的优劣,最后是面临的问题和突破方向。

图3,KcsA的结构(PDBid:1BL8)
| Exp.Method | Proteins | Nucleic Acids | Protein/NA Complexes | Other | Total |
| X-RAY | 80621 | 1502 | 4194 | 4 | 86321 |
| NMR | 9024 | 1072 | 197 | 7 | 10300 |
| ELECTRON MICROSCOPY | 508 | 51 | 170 | 0 | 729 |
| HYBRID | 57 | 3 | 2 | 1 | 63 |
| other | 155 | 4 | 6 | 13 | 178 |
| Total | 90365 | 2632 | 4569 | 25 | 97591 |
表1,PDB收集的结构统计
X射线其实是X射线晶体学的简写,一般使用的波长是1埃左右,也就是0.1nm。这个尺度刚好也是原子半径大小。
X射线也是一种光,我们眼睛能看到东西也是因为光。物体发射或者反射光进入我们的眼睛,我们通过对光子的响应知道了物体的存在。你如何知道一张纸的厚度?一般纸张的厚度在0.1mm左右,就是100微米,100,000nm。你经常见到的尺子的刻度是多少?1mm,显然你无法直接用尺子去量一张纸的厚度。需要一个跟测量的物体相一致的探针,这就是X光。我们可以用显微镜放大物体,看到细胞甚至是细胞器。但是可见光的波长是几百nm,是无法直接看到原子。
晶体学的意思是,我们使用的样品是晶体。这并不复杂,我们平时看到的食盐、雪花都是晶体,世俗中最有价值的晶体当然是钻石了,那是碳原子组成的晶体。如果你把木炭放在高温高压的环境下,辅以一定的条件,就能得到钻石了。最重要的是样品中原子的排列,在晶体中,原子或者分子按特定的顺序搭起来,排列的整整齐齐,就如军队中阅兵方阵。
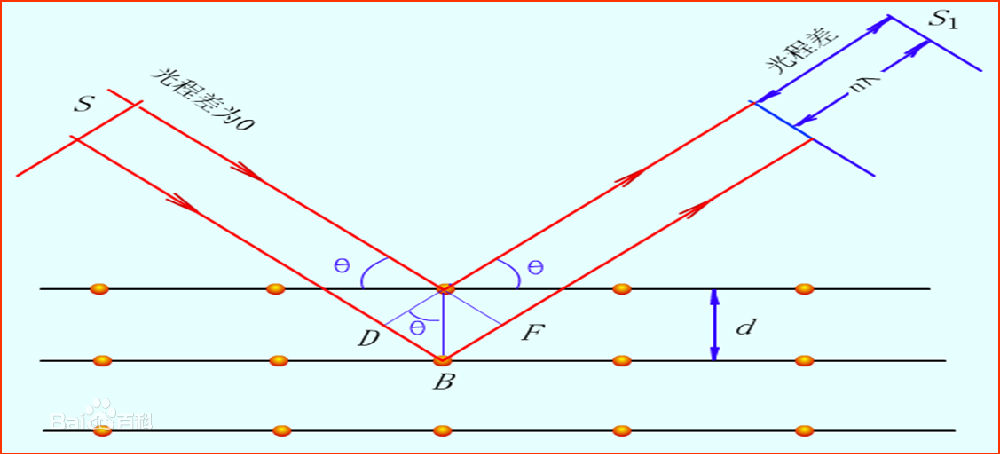
当X光照射到晶体上时,会引起散射。就是光波会激发原子发出同样频率的波,由于晶体中原子排列的有序性,在特定的方向上,散射的光得到了增强。图4展示了Bragg方程的几何关系。在两个晶面上都发生了反射,远场条件下,它们的光程差是,当这个光程差满足光波长的n倍时才产生衍射。因为晶体是无数这样的面的叠加,所以在这个方向上的光变得极强,三维条件下就是一个点。晶体学中还有一个跟Bragg方程等效的方程,劳厄方程,它从晶体的特性出发,构置倒晶格向量,只有当波矢是倒格矢的整数倍相加才能发生衍射。

图4,Bragg方程的导出
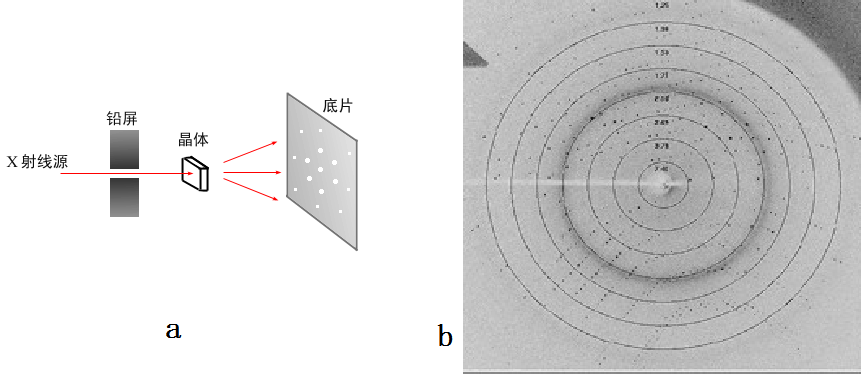
为了进行蛋白质结构测定,首先需要将蛋白质进行结晶,然后在X光源上进行衍射实验。衍射装置如图5a所示,一般晶体是装在一个旋转轴上,可以360°旋转。现在也很少用底片,比较常用的是成像板,同步辐射装置上一般使用CCD,这样读写速度更快,方便实验的进行。图5b给出的一张真正的蛋白质衍射点,图中的白线叫beamstop,就是挡住X射线,以免直接打到CCD上。X光照到晶体上,被散射的部分很少,余下的都直接穿过晶体,如果不挡住的话会把CCD烧坏的。想象一下CT,就知道X射线的穿透能力了。图b中你也会发现中间的点多一些,颜色也深一些。这就涉及到一个强度的问题。
一个电子对X光的弹性散射(Thompson散射),散射强度如下:
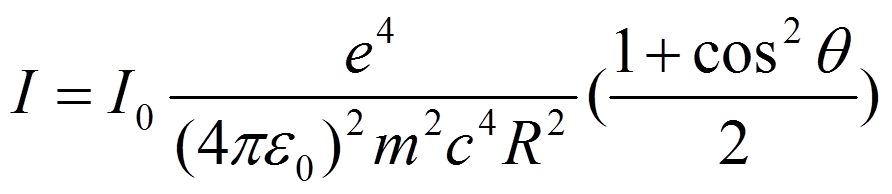 。
。
图5,a,衍射装置的几何结构;b,一张蛋白质晶体的衍射图
需要注意的跟质量的平方成反比,质子的质量是电子的1800倍,所以电子的散射强度比质子强得多,我们得到的信号主要来自于电子的散射。另外就是跟散射角度的关系,角度越高,散射越弱。当有多个电子的时候,电子之间的位置关系用来表示,波矢用来表示,散射出来的波就有一个相位关系,用电子密度来表示电子的位置,就有
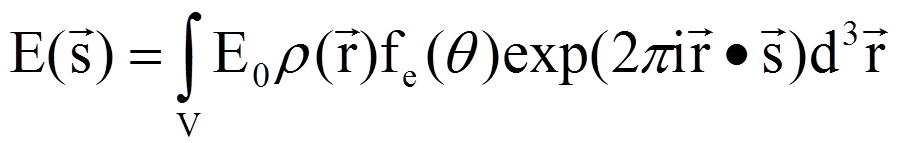
CCD测量的是这个值的平方,从这个关系也可以知道,衍射点的强度是电子密度的傅里叶变换。当测得这些点的衍射强度之后,通过反傅里叶变换就可以得到电子密度。这里面的问题是相位的丢失,当原子数目很少的时候,可以直观地猜出来原子的相对位置。原子数目很多的时候,相对位置的数目就是原子数的阶乘,血红蛋白有将近6000个原子,这个方法就失效了。所以人们想了很多办法来解决这个问题,比如同晶置换、反常散射。而且由于计算机技术的发展,解结构变成一个非常成熟的技术。所以你会看到每年有越来越多的结构被解析出来。
核磁共振是指核磁矩不为零的原子核,在外磁场的作用下,核自旋能级发生塞曼分裂,共振吸收某一特定频率的射频辐射的物理过程。原子核处于分子内部,分子中运动的电子受到外磁场的作用,产生感生电流。这一感生电流在核上产生感生磁场,感生磁场与外磁场相互叠加,使核上受到的有效场发生变化。我们把这一现象称为原子核受到了屏蔽。由于屏蔽作用使得同一种原子核由于所处的化学环境不同,核磁共振频率略有不同,这被称为化学位移,常以δ表示。屏蔽作用的大小与核外电子云密度有关,后者又与原子核的化学环境有关,因此可根据化学位移的大小,来考察原子核所处的化学环境,从而对化合物进行结构分析。
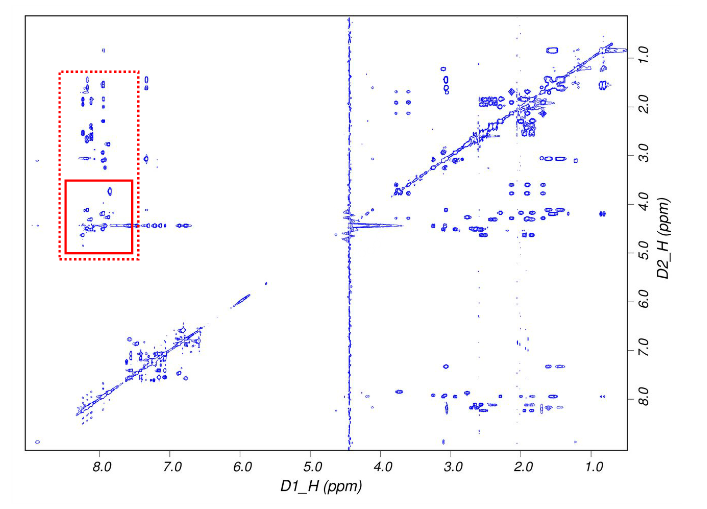
图6是一幅真实的二维核磁共振谱,里面的蓝色点代表该强度下的原子的化学位移信息。坐标轴ppm就是化学位移,通过使用同一参照样品使得其独立于外加磁场。我们所得到的是一系列这样的原子间距信息,但是无法得到全局的结构,这样就需要进行动力学优化。最后得到的结构模型就是一系列动力学最优解。
相对于晶体学,这种方法的局限性是不能测太大的蛋白质的结构,得到的结果也不如晶体学那样可靠,优点在于是在溶液中进行的,不像晶体学需要结晶,这样会使结构受到堆积力的作用,也会造成样品制造上的困难。

图6,二维核磁共振谱
电镜的全称是电子显微镜。使用的是电子作为探针,用电磁透镜代替了光学透镜并使用荧光屏将肉眼不可见电子束成像。为什么不使用透镜来让x光成像呢?原因在于硬X射线的穿透性,目前还无法做X射线透镜。

图7,a为扫描电子显微镜的构造图,b为天花病毒的结构
电子打在样品上,会激发出二次电子、或X射线等,通过探测二次电子可以得到当前位置的电子强度信息。然后移到附近,这样就可以得到图7b中的图像。图中的颜色是假彩色,真实的信息是二次电子的强弱。按照成像理论,这样得到的只是一个样品的截面,如果有许多全同的样品,扫描得到不同截面的信息,就可以利用计算机技术将这些截面组合起来,得到样品的三维结构。
这里面存在的问题是,电子的散射很强,很容易被空气散射,所以需要高真空环境。这样一来样品就不能是活的。由于技术原因,目前最好的分辨率不到3埃,这和晶体学相比是有差距的。
3、蛋白质结构测定的未来 前面大致介绍了一下蛋白质结构测定的常用方法。总体来说,这几种方法越来越成熟,每年测得的结构也越来越多。下表是截至2014年2月PDB数据库中的结构数目,可以看到结构数目呈指数增长。国内而言,也有越来越多的生物学家进入结构测定这一领域,因为结构确实能解释他们研究中的很多问题,加深对于生物意义的理解。

单纯就结构测定而言,能解释的问题是有限。一个蛋白长成一个样子,然后我们说这样就可以跟DNA结合,可以作为离子通道,可以跟某个小分子结合,这些在生物学实验中可能就已经得到验证了。给你一个蛋白结构,如果对于其在细胞中的定位以及功能了解很少的话,其实是很难讲一个好故事的。这就是生物学家做结构的优势。他们可能在细胞或者生化层面做研究,到了分子层面,以前的那些疑问可能都会得到解答。他原来就知道蛋白有某种特性,必须与DNA结合,拿到结构一看,蛋白质的凹槽正好可以结合DNA。某种疾病引起了基因突变,他把正常的和突变的蛋白结构一比较,就可以发现问题出现在什么地方。这是结构测定的一大前景。
前面已经说过电镜和NMR的问题,晶体学的最大瓶颈在于结晶。结晶的方法是得到高纯度的蛋白,然后将蛋白加入各种配好的溶液中进行结晶。具体哪个溶液能够长出晶体是不知道的,只能不停地去尝试。而且已经测得的结构如此之多,那些容易结晶的蛋白质可能已经被尝试过了,现在的方向是复合物。由好几个蛋白质通过相互作用构成的复合物一起实现一个功能,但是和单个蛋白相比,复合物之间的相互作用比较弱,这样对溶液环境的要求就更高。也就是说更难找到结晶条件了,而且就算是长成晶体,由于复合物结构复杂,很难是完美晶体,衍射的分辨率就上不去。
回到结构本身。在小分子领域,量子化学的计算是相当精确的。但在蛋白质结构中,由于原子众多,用量化计算的话计算量非常大,所以常用的分子动力学。通过构置分子力场,就是分子中原子相互作用规则,然后利用最优化方法,得到一个“能量”最小的结构。而为了兼顾精度和计算速度,人们会使用QM/MM这样的组合,即在核心区域使用量化进行计算,而在外围使用分子力学。蛋白质中的原子是作为一个整体存在的,配体结合在活性位点会影响整个蛋白的结构。很多蛋白使用ATP作为动力来源,ATP末端磷酸基团水解时,释放出的能量是30.54 kJ/mol。这样的能量够离子泵运作一次。显然,你结晶的时候是无法把ATP水解的过程固定住,NMR测定一个结构的时间要几天。而一个电子在蛋白质中的转移可能就在ps量级。这很好理解,一个反应中,中间态是不稳定,只有初态和末态才能大量存在,就如血红蛋白中结合氧和没有结合氧,你很难找到一个即将结合氧的结构。但是我们可以自己制造。
时间分辨的晶体学是在飞秒化学的基础上发展起来的。利用飞秒激光作为时间基准,可以在反应之后的特定时间进行测量,这样就可能得到反应中间态的信息。比如肌红蛋白结合氧的实验,但是氧和肌红蛋白结合的能力不如CO(CO中毒的原因就在于此,结合到血红蛋白之后不容易解离,造成氧输运受阻),所以一般这类实验采用的是结合CO的肌红蛋白,使用特定频率的光照,可以使CO脱离蛋白中的铁原子,脱离之后CO是不是飞出结合的口袋呢?可以在解离之后特定的时间去测量红外光谱,这样响应时间很快。而要直观地看到结构,则使用X射线,在150ps内,CO还是停留在肌红蛋白的口袋中,大约在1ms之内会重新结合到铁原子上。为了更快地进行测量,往往使用“白光”,这样的衍射叫劳厄衍射,一张衍射图就可能解出结构。显然这样的实验也存在问题,比如使用晶体,这样光解实验变化的区域只能活性位点附近。
如果有一种方法能够得到单分子的结构,那么就可以很方便地得到中间态。这就是X射线自由电子激光最诱人的应用。X射线自由电子激光(FEL)的基本原理是通过自由电子和辐射的相互作用,电子将能量传送给辐射而使辐射强度放大。由直线加速器产生的电子束流,经过几次压缩后将能产生超短的脉冲长度(fs量级),将为研究物质的微观动态结构以及非线性过程提供前所未有的机会。
图8是利用FEL进行单颗粒成像的示意图。蛋白颗粒从一个质谱仪中喷出,一束相干X光照射上去,由于光束其高的能量,蛋白被击碎。但是在被击碎之前,已经将X射线散射出来,里面含有蛋白结构信息。这仍然是电子密度的傅里叶变换,可以反向重建蛋白质的结构。现在的问题是一张图的信号太差,需要许多张一样的图平均来增强信号,得到的仍然是结构的一个截面,这与电镜类似,所以需要多张图来重建结构。最大的问题是,高角度的散射信号非常弱,不到一个光子,于是分辨率也上不去。
使用更强的光源,更短的脉冲,以及更好的探测器,是否有可能使用一张图就解出结构来呢?如果答案是能的话,那么要多强的光,多短的脉冲,多好的探测器?现在美国斯坦福的LCLS已经为用户进行供光,位于德国汉堡的X-FEL和日本的SCSS都在建设之中。相关的实验已经进行了几十个,但对于单张图成像方法和理论都需要更多的探索。
写到这里,其实很多东西没有写明白,只是给出一个大致的轮廓。在单分子测量领域,光镊和磁镊对分子力学进行了很多有意义的测量;荧光能量共振转移(FRET)已经成为检测活体中生物大分子纳米级距离和纳米级距离变化的有力工具,在生物大分子相互作用时间分析有着广泛的应用;而生物学家在生物大分子相互作用也有很多非常具有创造性的方法。总体来看,时间分辨的大分子结构测定只是我们对于分子的理解需要的更多信息的一种可能,但是是最直观的一种!

图8,单颗粒成像示意图
http://blog.sciencenet.cn/blog-502850-767838.html 此文来自科学网刘广峰博客,转载请注明出处。
3 苏光松 王守业 郑永军
该博文允许注册用户评论 请点击登录 评论 (2 个评论)
- [2]高友鹤
- 谢谢博主指点!
- [1]高友鹤
- 非常外行。一直没弄明白蛋白质在晶体时的结构和原来在细胞里的那个复杂环境下会一样吗?不同方法解析出来的结构一样吗?和体内原来的结构近似的程度有多少?也许没人能确切知道真正的结构?完全外行,瞎提问题。
- 博主回复(2014-2-15 21:25):你如果关心这些问题的话,理解我说的这几个方法都有答案。蛋白质在晶体时和细胞中不一样,不仅是细胞中的溶液环境复杂,蛋白结晶的时候会在一定程度上改变结构。不同的方法解析出来的结构不一样,但大致是一样,看完第一节就有答案。和体内的结构非常近似。什么是真正的结构?测量都是有误差的。
A physicist has proposed the provocative idea that life exists because the law of increasing entropy drives matter to acquire life-like physical properties

No comments:
Post a Comment